![[mllib] Pyspark Kmeans 알고리즘 사용법](https://img1.daumcdn.net/thumb/R750x0/?scode=mtistory2&fname=https%3A%2F%2Fblog.kakaocdn.net%2Fdn%2FtzEI7%2FbtryEyqO1h2%2FMT5hZp7bKOHAWNqUzHP8F0%2Fimg.png)

Pyspark Mllib에서 Kmeans 알고리즘 사용법을 다루는 글이다. (데이터브릭스 이용)

1. 라이브러리 세팅
import pandas as pd
# 스파크 sql 내장함수
from pyspark.sql import *
# 스파크 sql 내 자료구조 타입
from pyspark.sql.types import *
# 스파크 sql 내 여러 함수들
from pyspark.sql.functions import *
# 스케일러, VectorAssembler -> 여러 자료값들을 하나의 vector로 모아줌
from pyspark.ml.feature import MinMaxScaler, VectorAssembler
# mllib에서 연산을 위해서는 vector형 자료 구조로 변환
from pyspark.ml.linalg import Vectors
from pyspark.ml.clustering import KMeans
# 실루엣 계수
from pyspark.ml.evaluation import ClusteringEvaluator
각각 필요한 라이브러리들을 정리해봤다. 데이터브릭스 14일 free_trial 버전을 이용하여 본 코드를 실행중인데, 데이터브릭스의 경우 SparkSession이 자동으로 생성되어 있기 때문에 SparkSession을 build하는 부분은 생략한다.
SparkSession 관련된 부분은 공홈 참조
https://spark.apache.org/docs/latest/sql-getting-started.html
Getting Started - Spark 3.2.1 Documentation
spark.apache.org
2. 데이터 세팅
데이터는 sklearn 내장 데이터인 캘리포니아 집값 데이터를 이용할 것이다. 아래와 같이 데이터를 불러오고
data_url = "http://lib.stat.cmu.edu/datasets/boston"
raw_df = pd.read_csv(data_url, sep="\s+", skiprows=22, header=None)
RDD 데이터 프레임으로 변환해준다.
spark_df = spark.createDataFrame(raw_df)
아웃풋을 살펴보니 null값이 존재한다.
spark_df.show(5)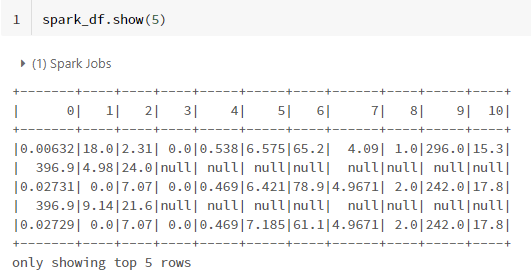
알고리즘을 실행해보는 것이 목적이므로, 귀찮으니 다 0으로 때려박는다.
spark_df_v2 = spark_df.fillna(0)
spark_df_v2.show(5)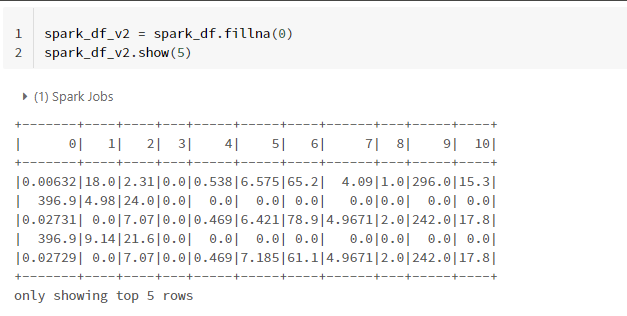
-----------깰끔----------------
3. Mllib 사용을 위한 데이터 전처리
3-1. dense vector만들기
mllib사용을 위해서는 데이터를 vector 형태로 변환해주어야 한다.
from pyspark.ml.linalg import Vectors이를위해서 Vectors를 따로 설치해주어야 하는데
https://spark.apache.org/docs/latest/api/python/reference/api/pyspark.ml.linalg.Vectors.html
Vectors — PySpark 3.2.1 documentation
Create a sparse vector, using either a dictionary, a list of (index, value) pairs, or two separate arrays of indices and values (sorted by index).
spark.apache.org
Dense vectors are simply represented as NumPy array objects, so there is no need to covert them for use in MLlib. For sparse vectors, the factory methods in this class create an MLlib-compatible type, or users can pass in SciPy’s scipy.sparse column vectors.
공홈 설명만 보면 numpy array와 유사한 자료구조를 mllib에도 만들어둔 것 같다. 적힌 말만 보면 numpy array도 array에서 사용가능한건가? 나중에 테스트 해봐야겠다.
어쨌든, VectorAssembler를 이용해서 기존 RDD 데이터프레임 내부에 있는 컬럼들의 값들을 하나의 dense vector로 합쳐준다.
Assembler = VectorAssembler(inputCols = spark_df_v2.columns, outputCol='features')
mllib_dataset = Assembler.transform(spark_df_v2)
뷰로 만들어서 살펴보면 좀 더 이 자료구조에 대해서 쉽게 이해할 수 있다.
mllib_dataset.createOrReplaceTempView("mllib_dataset")
참고로 %위에 언어를 적어주면 해당 언어를 사용할 수 있다.
%sql
select *
from mllib_dataset
살펴보면 values에 [] numpy array와 유사한 형태로 값들이 담겨있다. 행별로 array 사이즈가 다른데, 아마 0이 아닌 값들의 index만 indices에 따로 남기고 values에도 0이 아닌 값들만 남기는 것 같다.
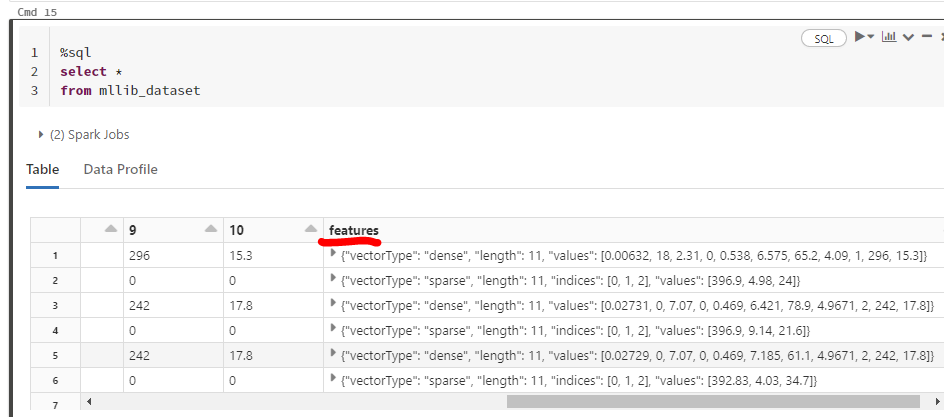
3-2. 스케일링하기
kmeans는 거리계산 기반 알고리즘이기 때문에 스케일에 영향을 받을 수 있어, 스케일링 작업이 필수다.
minMax = MinMaxScaler().setMin(0).setMax(1).setInputCol('features')
minMax_fit = minMax.fit(mllib_dataset)
minMax_transform = minMax_fit.transform(mllib_dataset)
setMin과 setMax를 이용해 min, max 범위를 정할 수 있다. 최종 output은 minMax_transform에 저장되는데, 이번에도 view를 만들어서 살펴보자.
minMax_transform.createOrReplaceTempView("minmax_output")%sql
select *
from minmax_output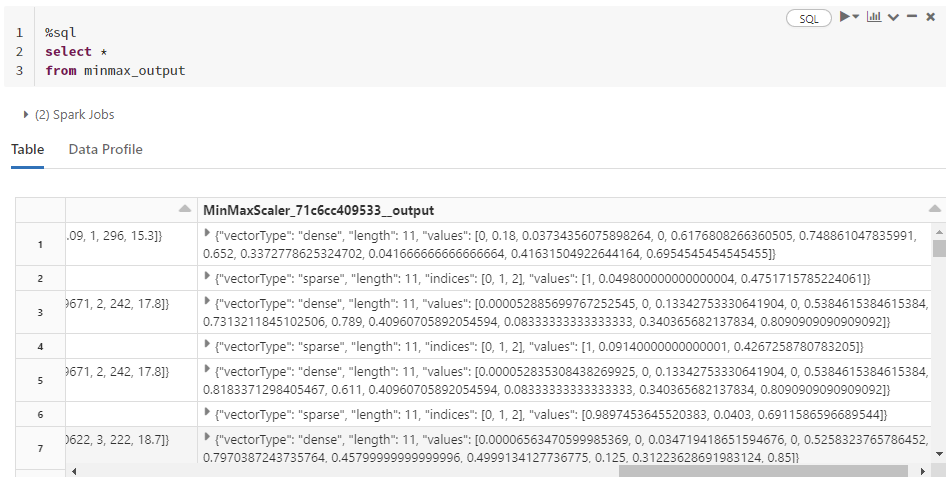
아까와의 차이점으로, values에 있는 값들이 0~1사이로 스케일링 된 것을 알 수 있다. 여기까지 하면 전처리도 끝이다. 알고리즘 실행이 목적이기 때문에 다른 추가작업은 하지 않았다.
4. Kmeans 실행
km = KMeans(featuresCol='features').setK(5)
km_fit = km.fit(minMax_transform)
cluster_label = km_fit.transform(minMax_transform)
클러스터링 결과는 cluster_label안에 저장된다.
cluster_label.select(col('prediction')).show(5)
실루엣 계수도 확인해볼 수 있다.
evaluator = ClusteringEvaluator()
silhouette_score = evaluator.evaluate(cluster_label)
silhouette_score
이상 지금까지 mllib에서 clustering 알고리즘을 실행해봤다. 손에 익어서 그런지, sklearn이 더 편한 것 같으면서도 과정만 생각해보면 sklearn과 큰 차이는 없는 것 같다.
'딥상어동의 딥한 프로그래밍 > Spark' 카테고리의 다른 글
| [Spark] map, 그런데 flat을 곁들인 - flatMap (0) | 2022.08.28 |
|---|---|
| [PySpark] 자료 구조와 연산 원리 - 스파크 누구냐 넌? (4) | 2022.06.28 |
| [Pyspark] from pyspark.sql import * VS from pyspark.sql.functions import * (0) | 2022.04.07 |
| [PySpark] Python 내장 함수 사용시 발생하는 오류 (0) | 2021.07.21 |
| 스파크의 실행 계획 (0) | 2021.04.21 |

제 블로그에 와주셔서 감사합니다! 다들 오늘 하루도 좋은 일 있으시길~~
포스팅이 좋았다면 "좋아요❤️" 또는 "구독👍🏻" 해주세요!
![[Spark] map, 그런데 flat을 곁들인 - flatMap](https://img1.daumcdn.net/thumb/R750x0/?scode=mtistory2&fname=https%3A%2F%2Fblog.kakaocdn.net%2Fdn%2FcdId0h%2FbtrKpP1irHZ%2Ftb1QJJTM3rEjfdQnINkzvK%2Fimg.png)
![[PySpark] 자료 구조와 연산 원리 - 스파크 누구냐 넌?](https://img1.daumcdn.net/thumb/R750x0/?scode=mtistory2&fname=https%3A%2F%2Fblog.kakaocdn.net%2Fdn%2FbjIGfE%2FbtrFIc7jjiO%2FGC0CEDGCAuxW1gqbthEKrk%2Fimg.png)
![[Pyspark] from pyspark.sql import * VS from pyspark.sql.functions import *](https://img1.daumcdn.net/thumb/R750x0/?scode=mtistory2&fname=https%3A%2F%2Fblog.kakaocdn.net%2Fdn%2Fbu0YXE%2FbtryE2ZuPLs%2FnM8YAOQ7cu7HBZl77lhaU1%2Fimg.png)